{
"cells": [
{
"cell_type": "markdown",
"metadata": {},
"source": [
"\n",
"\n",
"*(C) Copyright Franck CHEVRIER 2019-2020 http://www.python-lycee.com/*\n",
"\n",
" Pour exécuter une saisie Python, sélectionner la cellule et valider avec SHIFT+Entrée."
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"# ADN, ARN et protéines\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"Le but de cette activité est de simuler la transcription de brins d'ADN en ARN et la traduction de l'ARN en protéines.\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"## 1. Génération d'une chaîne d'ADN\n",
"\n",
"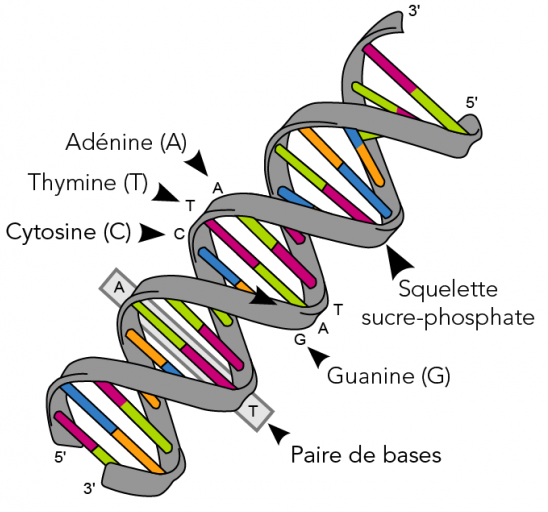"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 1.1. Compléter la structure suivante, contenant la liste des nucléotides pouvant composer un brin d'ADN. \n",
"\n",
"Attention : Penser ensuite à exécuter la zone ci-dessous (et les suivantes) avec SHIFT+Entrée."
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Définition de la liste des nucléotides de l'ADN\n",
"#(A COMPLETER)\n",
"Nucleotide_ADN=['A','T']"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 1.2. La fonction ADN_Aleatoire ci-dessous permet de générer une chaîne ADN de 3n nucléotides. Effectuer la saisie permettant de créer une chaîne nommée ADN, composée de 30 nucléotides, et afficher cette chaîne. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#import du module random pour la génération aléatoire de l'ADN\n",
"from random import*\n",
"\n",
"def ADN_Aleatoire(n):\n",
" \"Fonction qui crée un brin d'ADN de longueur 3*n\"\n",
" #Création d'une chaîne vide\n",
" ADN='' \n",
" for k in range(3*n):\n",
" #Ajouts successifs de nucléotides choisis au hasard\n",
" ADN += choice(Nucleotide_ADN)\n",
" return ADN"
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Effectuer la saisie\n",
"\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 1.3. Compléter la structure suivante, qui définit la correspondance des nucléotides entre 2 brins d'ADN correspondants.. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Définition de la correspondance des nucléotides des brins d'ADN \n",
"#(A COMPLETER)\n",
"Brin_correspondant={ 'A':'T' , 'T':'A' }"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 1.4. La fonction Creer_deuxieme_brin ci-dessous permet de générer le brin correspondant d'un brin d'ADN donné. Effectuer la saisie permettant de créer le brin ADN2, correspondant au brin ADN précédemment généré, et afficher le résultat. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"def Creer_deuxieme_brin(Brin_ADN):\n",
" \"Fonction qui reçoit un brin d'ADN et renvoie le 2e brin correspondant\"\n",
" Deuxieme_brin=''\n",
" for nucleotide in Brin_ADN:\n",
" Deuxieme_brin += Brin_correspondant[nucleotide]\n",
" return Deuxieme_brin"
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Effectuer la saisie\n",
"\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"## 2. Transcription de l'ADN en ARN\n",
"\n",
"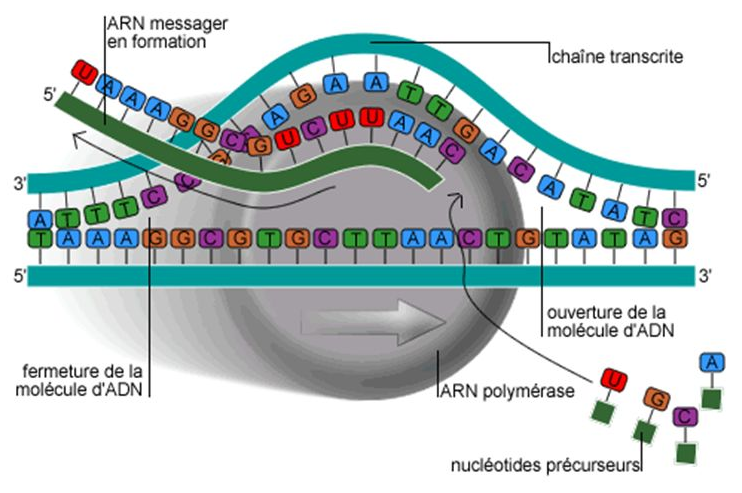"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 2.1. Compléter la structure suivante, qui définit la correspondance des nucléotides de l'ADN et des nucléotides de l'ARN. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Définition de la correspondance des nucléotides de l'ADN vers ARN\n",
"#(A COMPLETER)\n",
"ARN_correspondant={ 'A':'U' , 'C':'G' }"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 2.2. La fonction Transcription ci-dessous permet de transcrire en ARN un brin d'ADN donné. Effectuer la saisie permettant de créer le brin ARN, correspondant au brin ADN précédemment généré, et afficher le résultat. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"def Transcription(Brin_ADN):\n",
" \"Fonction qui réalise la transcription d'un brin d'ADN en chaîne ARN\"\n",
" ARN=''\n",
" for nucleotide in Brin_ADN:\n",
" ARN += ARN_correspondant[nucleotide]\n",
" return ARN"
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Effectuer la saisie\n",
"\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"## 3. Traduction de l'ARN\n",
"\n",
"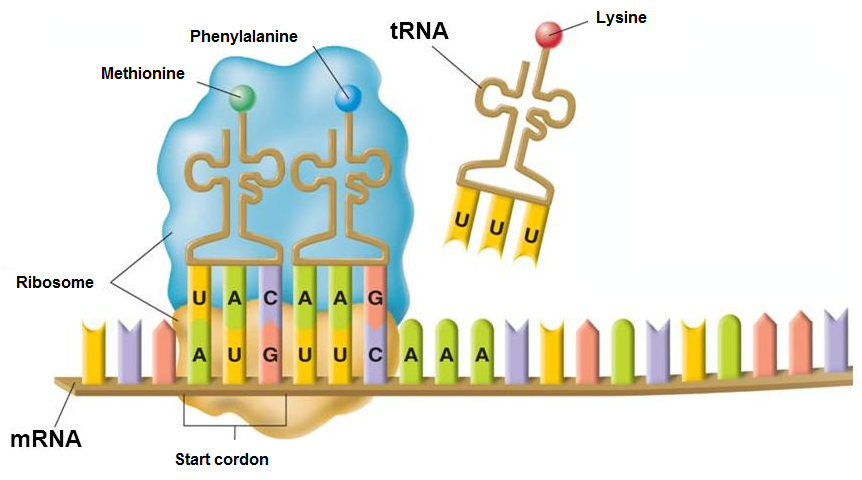"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
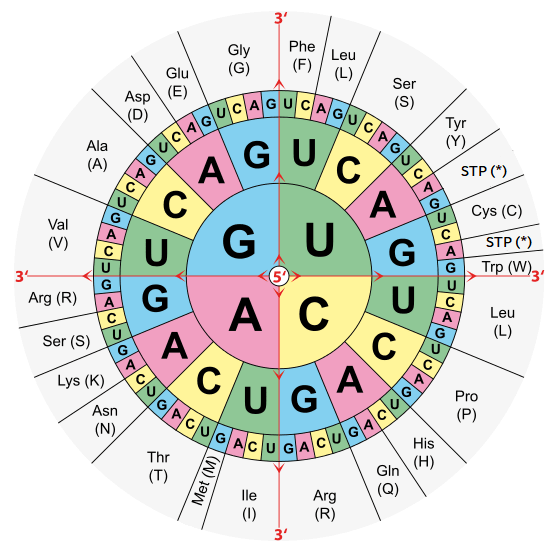
" 3.1. Compléter la structure suivante, qui définit la correspondance des nucléotides de l'ADN et des nucléotides de l'ARN. \n",
"\n",
"On pourra s'aider du schéma ci-dessous et on codera 'STP' pour l'arrêt.. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Définition de la correspondance des acides aminés et des triplets de nucléotides:\n",
"#(A COMPLETER)\n",
"Acides_amines={ 'His': ['CAU','CAC'] , 'Gln': ['CAA','CAG'] , 'Pro': ['CCU','CCC','CCA','CCG'] }"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
""
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 3.2. La fonction Acide_amine_correspondant ci-dessous réalise la traduction d'un triplet de nucléotides en acide aminé, et la fonction Traduction réalise la traduction d'une chaîne ARN en protéine.\n",
"\n",
" Effectuer la saisie nécessaire pour créer la protéine, nommée Proteine, obtenue avec le brin ARN précédemment généré."
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"def Acide_amine_correspondant(Triplet):\n",
" \"Fonction qui réalise la traduction d'un triplet de nucléotide en acide aminé\"\n",
" for Ac_am in Acides_amines:\n",
" for Triplet_correspondant in Acides_amines[Ac_am]:\n",
" if Triplet==Triplet_correspondant:\n",
" return Ac_am\n",
" return False\n",
"\n",
"def Traduction(ARN):\n",
" \"Fonction qui réalise la traduction d'une chaîne ARN en Proteine\"\n",
" Proteine=[]\n",
" for k in range(0,len(ARN)-2,3):\n",
" Ac_am=Acide_amine_correspondant(ARN[k:k+3])\n",
" if Ac_am=='STP': break\n",
" else:\n",
" Proteine.append(Ac_am)\n",
" return Proteine"
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Effectuer la saisie\n",
"\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 3.3. Compléter la structure suivante, qui définit la correspondance des acides aminés et de leurs nomenclatures simplifiées.. \n",
"\n",
"Ces nomenclatures sont précisées sur le schéma précédent.. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"Code_Ac_am={ 'His': 'H' , 'Gln': 'Q' , 'Pro': 'P' }\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
" 3.4. La fonction Simplification ci-dessous permet la simplification d'une liste d'acides aminés. Effectuer la saisie nécessaire pour simplifier Proteine précédemment généré. "
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"def Simplification(Proteine):\n",
" \"fonction qui renvoie une protéine sous sa forme simplifiée\"\n",
" Proteine_simp=''\n",
" for Ac_am in Proteine:\n",
" Proteine_simp += Code_Ac_am[Ac_am]\n",
" return Proteine_simp\n"
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"#Effectuer la saisie\n",
"\n"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"## 4. Annexe : Vérification de cohérence."
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"Exécuter le programme ci-dessous, pour obtenir une vérification de la cohérence de votre saisie de la correspondance des acides aminés avec les triplets de nucléotides."
]
},
{
"cell_type": "code",
"execution_count": null,
"metadata": {},
"outputs": [],
"source": [
"Nucleotide_ARN=[ARN_correspondant[nucleotide] for nucleotide in Nucleotide_ADN]\n",
"\n",
"def Verif_correspondance_acides_amines():\n",
" \"\"\"\n",
" Complément:\n",
" fonction de vérification de la correspondance\n",
" entre Acides aminés et triplets\n",
" \"\"\"\n",
" \n",
" print(\"\\nRECAPITULATIF DES SAISIES:\")\n",
" \n",
" compteur_triplets=0\n",
" for ac_am in Acides_amines:\n",
" print(ac_am, [Triplet for Triplet in Acides_amines[ac_am]])\n",
" compteur_triplets += len(Acides_amines[ac_am])\n",
" print(\" Nombres d'acides aminés écrits\",len(Acides_amines))\n",
" print(\" Nombres de triplets écrits (y compris STP):\",compteur_triplets)\n",
" \n",
" print(\"\\nVERIFICATION DES SAISIES:\")\n",
" \n",
" Liste_oublis=[]\n",
" \n",
" for n1 in Nucleotide_ARN:\n",
" for n2 in Nucleotide_ARN:\n",
" for n3 in Nucleotide_ARN:\n",
" Triplet=n1+n2+n3\n",
" if not Acide_amine_correspondant(Triplet):\n",
" Liste_oublis.append(Triplet)\n",
" \n",
" nbre_oublis=len(Liste_oublis)\n",
" print(\"Il y a\",nbre_oublis,\"triplet\"+(\"s\" if nbre_oublis>1 else \"\"),\"non attribué\"+(\"s\" if nbre_oublis>1 else \"\"),\"\" if nbre_oublis==0 else \"qui est:\" if nbre_oublis==1 else \"qui sont:\")\n",
" for oubli in Liste_oublis: print(oubli)\n",
"\n",
" return None\n",
"\n",
"Verif_correspondance_acides_amines()"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"*(C) Copyright Franck CHEVRIER 2019-2020 http://www.python-lycee.com/*"
]
},
{
"cell_type": "markdown",
"metadata": {},
"source": [
"*Remerciements particuliers à Christine DEUTSCHMANN pour son aide précieuse.*"
]
}
],
"metadata": {
"kernelspec": {
"display_name": "Python 3",
"language": "python",
"name": "python3"
},
"language_info": {
"codemirror_mode": {
"name": "ipython",
"version": 3
},
"file_extension": ".py",
"mimetype": "text/x-python",
"name": "python",
"nbconvert_exporter": "python",
"pygments_lexer": "ipython3",
"version": "3.6.4"
}
},
"nbformat": 4,
"nbformat_minor": 2
}