---
title: "Modelos de regressão"
subtitle: "Aula 6"
author: "Bruno Montezano"
institute: "Grupo Alliance
Programa de Pós-Graduação em Psiquiatria e Ciências do Comportamento
Universidade Federal do Rio Grande do Sul"
date: last-modified
date-format: long
lang: pt-br
execute:
echo: true
format:
revealjs:
incremental: true
smaller: true
theme: [default, ../assets/custom.scss]
logo: "../assets/logo_ufrgs.png"
---
## Conteúdo de hoje
- Conceito
- Regressão linear simples
- Regressão linear múltipla
- Coeficiente de determinação
- Preditores categóricos
- Desafios
## Ciclo da ciência de dados
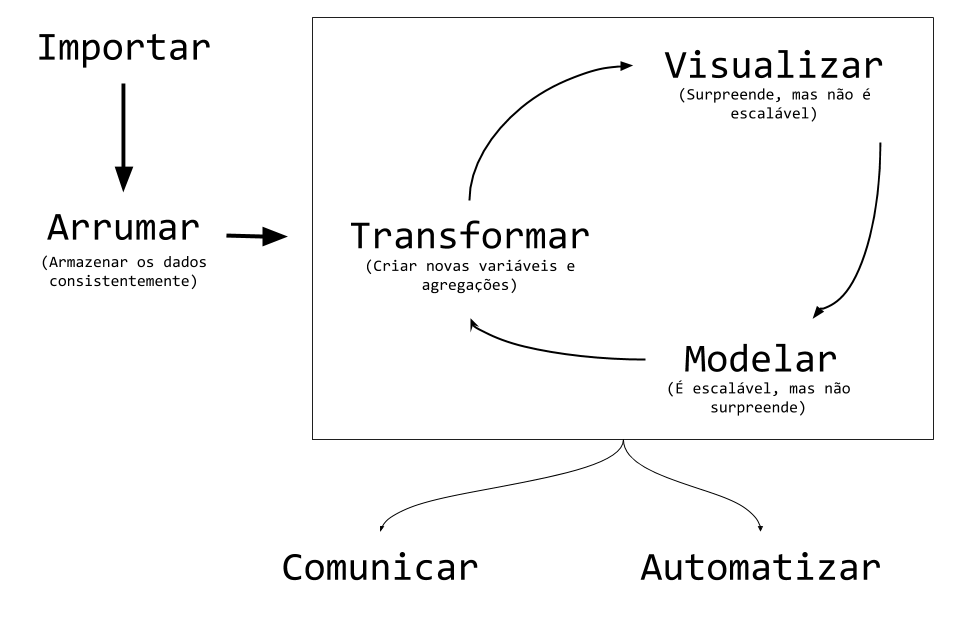{fig-align="center" width=85%}
## Regressão linear
* Método estatístico para sumarizar e estudar relações entre duas variáveis
contínuas
* $X$: preditor ou variável explicativa ou variável independente
* $Y$: desfecho ou variável resposta ou variável dependente
* Exemplo: salário ($Y$) e anos de estudo ($X$)
. . .
```{r altura-peso-scatter}
#| echo: false
#| fig-align: "center"
#| out.width: "70%"
set.seed(1)
library(ggplot2)
altura <- round(runif(10, 150, 195), 1)
peso <- 0.5 * altura + rnorm(10, mean = 0, sd = 3)
medidas <- data.frame(altura, peso)
modelo <- lm(peso ~ altura,
data = medidas)
medidas$predito <- predict(modelo)
medidas$residuos <- medidas$peso - medidas$predito
ggplot(medidas,
aes(x = altura, y = peso)) +
geom_point(size = 4, color = "slateblue4") +
scale_x_continuous(labels = \(x) paste0(as.character(x), " cm")) +
scale_y_continuous(labels = \(x) paste0(as.character(x), " kg")) +
geom_smooth(method = "lm",
color = "grey20",
se = FALSE,
fullrange = TRUE) +
labs(x = "Altura",
y = "Peso") +
theme_minimal(base_size = 16,
base_family = "Charter")
```
## Qual a utilidade das retas?
```{r exemplos-retas}
#| echo: false
#| fig-align: "center"
#| out.width: "100%"
library(patchwork)
library(dplyr)
neg <- openintro::simulated_scatter |> filter(group == 1)
pos <- openintro::simulated_scatter |> filter(group == 2)
ran <- openintro::simulated_scatter |> filter(group == 3)
bad <- openintro::simulated_scatter |> filter(group == 5)
bad_reg <- bad |>
mutate(y = max(y) + min(y) - y) |>
ggplot(aes(x = x, y = y)) +
geom_point(size = 2,
alpha = 0.8,
color = "slateblue4") +
geom_smooth(method = "lm",
se = FALSE,
color = "gray20") +
labs(x = NULL,
y = NULL) +
theme_minimal(base_size = 16, base_family = "Charter")
p_neg <- ggplot(neg, aes(x = x, y = y)) +
geom_point(size = 2, alpha = 0.8, color = "slateblue4") +
geom_smooth(method = "lm", se = FALSE, color = "gray20") +
labs(x = NULL, y = NULL) +
theme_minimal(base_size = 16, base_family = "Charter")
p_pos <- ggplot(pos, aes(x = x, y = y)) +
geom_point(size = 2, alpha = 0.8, color = "slateblue4") +
geom_smooth(method = "lm", se = FALSE, color = "gray20") +
labs(x = NULL, y = NULL) +
theme_minimal(base_size = 16, base_family = "Charter")
p_ran <- ggplot(ran, aes(x = x, y = y)) +
geom_point(size = 2, alpha = 0.8, color = "slateblue4") +
geom_smooth(method = "lm", se = FALSE, color = "gray20") +
labs(x = NULL, y = NULL) +
theme_minimal(base_size = 16, base_family = "Charter")
p_neg + p_pos + p_ran + bad_reg +
plot_annotation(title = "Um modelo linear pode ser útil mesmo quando os pontos não passam diretamente pela reta.",
subtitle = "Em alguns casos, não será o método mais adequado.",
caption = "Adaptado do livro Introduction to Modern Statistics (Çetinkaya-Rundel e Hardin, 2023).",
theme = theme(text = element_text(family = "Charter")))
```
## Como a reta é construída?
Construída a partir da fórmula:
$Y = \beta_0 + \beta_1x_1 + e$ $\color{red}\longrightarrow$ $peso = -4.47 + 0.527 \times altura$
* $\beta_0$: intercepto, ou valor de $Y$ quando $X = 0$
* $\beta_1$: coeficiente angular (*slope*)
- Entendido como a mudança no $Y$ a partir do aumento de 1 unidade
no $x_1$
. . .
```{r altura-peso-ilustracao-equacao}
#| echo: false
#| fig-align: "center"
#| out.width: "70%"
ggplot(medidas,
aes(x = altura, y = peso)) +
geom_point(size = 2.5, color = "slateblue4",
alpha = 0.9) +
scale_x_continuous(labels = \(x) paste0(as.character(x), " cm"),
limits = c(0, 195)) +
scale_y_continuous(labels = \(x) paste0(as.character(x), " kg")) +
geom_smooth(method = "lm",
color = "grey20",
se = FALSE,
fullrange = TRUE) +
geom_point(aes(x = x, y = y),
data.frame(x = 0, y = -4.47),
size = 5,
color = "indianred1") +
labs(x = "Altura",
y = "Peso") +
geom_segment(x = 150, xend = 150, y = 75, yend = 0,
color = "tan1",
linewidth = 1.5) +
geom_segment(x = 150, xend = 0, y = 75, yend = 75,
color = "tan1",
linewidth = 1.5) +
geom_point(x = 150, y = 74.58,
size = 5,
color = "maroon1") +
theme_minimal(base_size = 16,
base_family = "Charter")
```
## Definições e nomenclaturas
* $x_1, x_2, \cdots, x_p$: variáveis explicativas (variáveis independentes ou
*features* ou preditores)
* $X = x_1, x_2, \cdots, x_p$: conjunto de todos os preditores
* $Y$: variável resposta (ou variável dependente ou *target*)
* $\hat{Y}$: valor esperado (ou predição ou valor estimado ou *fitted*)
* $f(X)$ pode ser entendida como "modelo" ou "hipótese"
. . .
No primeiro exemplo:
* $x_1$: `altura` --- Altura em centímetros
* $Y$: `peso` --- Peso em quilogramas
## Observado *vs.* Esperado
* $Y$ é um valor observado (ou verdade) a partir dos dados
* $\hat{Y}$ é um valor esperado (predição, ou valor estimado, ou *fitted*)
* $Y - \hat{Y}$ é o resíduo (ou erro)
. . .
Por definição, $\hat{Y} = f(X)$ que é o valor que a função $f$ retorna.
Em suma, nosso modelo.
```{r altura-peso-erro}
#| echo: false
#| fig-align: "center"
#| out.width: "60%"
ggplot(medidas,
aes(x = altura, y = peso)) +
geom_segment(
aes(
x = altura,
y = peso,
xend = altura,
yend = predito
),
color = "chocolate1",
linewidth = 1.5,
alpha = 0.8,
data = medidas
) +
geom_point(size = 3.5, color = "slateblue4") +
scale_x_continuous(labels = \(x) paste0(as.character(x), " cm")) +
scale_y_continuous(labels = \(x) paste0(as.character(x), " kg")) +
geom_smooth(method = "lm",
color = "grey20",
se = FALSE,
fullrange = TRUE) +
labs(x = "Altura",
y = "Peso") +
theme_minimal(base_size = 16,
base_family = "Charter")
```
## Por que ajustar uma $f$?
::: columns
::: {.column width="50%"}
### Predição
* Em muitas situações, $X$ está disponível facilmente mas $Y$ não é fácil de
descobrir (ou mesmo não é possível descobrí-lo).
* Queremos que $\hat{Y}=f(X)$ seja uma
boa estimativa (preveja bem o futuro).
* Exemplo: Com quantos gramas irá nascer um bebê na maternidade do HCPA?
:::
::: {.column width="50%"}
### Inferência
* Em inferência estamos mais interessados em entender a relação entre as variáveis explicativas $X$ e a variável resposta $Y$.
* Exemplos:
- Qual o efeito da idade na função cognitiva dos pacientes bipolares?
- Os níveis de estresse podem explicar a severidade de sintomas depressivos?
* Nessa aula, nosso foco é **inferência**.
:::
:::
## Vamos trabalhar com `gambás`
::: columns
::: {.column width="65%"}
Os dados apresentam informações de gambás da Austrália e Nova Guiné.
```{r ler-e-mostrar-gambas}
library(readr)
gambas <- read_csv("../dados/gambas.csv")
glimpse(gambas)
```
:::
::: {.column width="35%"}
{fig-align="center" width=85%}
:::
:::
## Exemplo com `gambás`
::: columns
::: {.column width="50%"}
#### Regressão linear simples
$y = \beta_0 + \beta_1x$
#### Exemplo
$cabeça = \beta_0 + \beta_1total$
```{r lm-cabeca-comprimento-total}
#| eval: false
# No R
lm(comprimento_cabeca ~ comprimento_total,
data = gambas)
```
:::
::: {.column width="50%"}
::: {.panel-tabset}
## Gráfico
```{r scatterplot-gambas}
#| echo: false
ggplot(gambas) +
geom_point(aes(x = comprimento_total, y = comprimento_cabeca),
size = 4,
alpha = 0.7) +
labs(x = "Comprimento total (em cm)",
y = "Comprimento da cabeça (em mm)") +
theme_minimal(18, "Charter")
```
## Reta na mão
```{r reta-na-mao-gambas}
#| echo: false
ggplot(gambas, aes(x = comprimento_total, y = comprimento_cabeca)) +
geom_point(size = 4,
alpha = 0.7) +
#geom_smooth(method = "lm",
# se = FALSE,
# color = "firebrick1",
# size = 2) +
geom_abline(intercept = 36,
slope = 0.7,
color = "dodgerblue4",
size = 2) +
labs(x = "Comprimento total (em cm)",
y = "Comprimento da cabeça (em mm)") +
theme_minimal(18, "Charter")
```
## Melhor reta
```{r melhor-reta-gambas}
#| echo: false
ggplot(gambas, aes(x = comprimento_total, y = comprimento_cabeca)) +
geom_point(size = 4,
alpha = 0.7) +
geom_smooth(method = "lm",
se = FALSE,
color = "firebrick1",
size = 2) +
geom_abline(intercept = 36,
slope = 0.7,
color = "dodgerblue4",
alpha = 0.6,
size = 2) +
labs(x = "Comprimento total (em cm)",
y = "Comprimento da cabeça (em mm)") +
theme_minimal(18, "Charter")
```
:::
:::
:::
## Melhor reta no R
$comprimento \,da \,cabeça = \beta_0 + \beta_1comprimento \,total$
. . .
```{r regressao-linear-gamba}
melhor_reta <- lm(comprimento_cabeca ~ comprimento_total, data = gambas)
melhor_reta
```
. . .
$comprimento \,da \,cabeça = 42.7 + 0.57 \times comprimento \,total$
. . .
Como a melhor reta é escolhida?
## "Melhor reta" baseado em que?
A gente quer a reta que *erre menos*.
. . .
Exemplo de medida de erro: **E**rro **Q**uadrático **M**édio.
$$
EQM = \sqrt{\frac{1}{N}\sum(y_i - \hat{y_i})^2}
$$
```{r comparar erro-reta-na-mao-e-melhor}
#| echo: false
#| fign-align: "center"
#| out.width: "70%"
set.seed(1)
melhor_reta <- lm(comprimento_cabeca ~ comprimento_total, data = gambas)
gambas_com_preds <- melhor_reta |>
broom::augment() |>
rename(pred_melhor_reta = .fitted) |>
mutate(
pred_reta_a_mao = 36 + 0.7 * comprimento_total
)
grafico_residuos_melhor_reta <- gambas_com_preds |>
ggplot(aes(x = comprimento_total, y = comprimento_cabeca)) +
geom_point(size = 2) +
geom_abline(
intercept = melhor_reta$coefficients[1],
slope = melhor_reta$coefficients[2],
size = 1,
colour = "salmon"
) +
geom_segment(aes(xend = comprimento_total, yend = pred_melhor_reta), colour = "purple", size = 0.8) +
labs(
subtitle = "Resíduos da Melhor Reta",
y = "Comprimento da cabeça (em mm)",
x = "Comprimento total (em cm)"
) +
theme_minimal(15, "Charter")
grafico_residuos_reta_a_mao <- gambas_com_preds |>
ggplot(aes(x = comprimento_total, y = comprimento_cabeca)) +
geom_point(size = 2) +
geom_abline(
intercept = 36,
slope = 0.7,
size = 1,
colour = "orange"
) +
geom_segment(aes(xend = comprimento_total, yend = pred_reta_a_mao), colour = "purple", size = 0.8) +
labs(
subtitle = "Resíduos da Reta Escolhida a Mão",
y = "Comprimento da cabeça (em mm)",
x = "Comprimento total (em cm)"
) +
theme_minimal(15, "Charter")
grafico_residuos_melhor_reta + grafico_residuos_reta_a_mao +
plot_annotation(title = "Os segmentos roxos são os resíduos (ou o quanto o modelo errou naqueles pontos).",
theme = theme_minimal(15, "Charter"))
```
## E se quisermos usar mais preditores?
::: columns
::: {.column width="50%"}
#### Regressão linear múltipla
$y = \beta_0 + \beta_1x_1 + \cdots + \beta_px_p$
#### Exemplo
$cabeça = \beta_0 + \beta_1total + \beta_2cauda$
```{r linear-multipla-no-r-gambas}
#| eval: false
# No R
lm(comprimento_cabeca ~ comprimento_total + comprimento_cauda,
data = gambas)
```
:::
::: {.column width="50%"}
```{r multipla-plotly}
#| echo: false
#| fig-align: "center"
#| out.width: "100%"
set.seed(1)
library(plotly)
x <- gambas$comprimento_total
y <- gambas$comprimento_cauda
z <- gambas$comprimento_cabeca
fit <- lm(z ~ x + y)
grid.lines <- 26
x.pred <- seq(min(x), max(x), length.out = grid.lines)
y.pred <- seq(min(y), max(y), length.out = grid.lines)
xy <- expand.grid(x = x.pred, y = y.pred)
z.pred <- matrix(predict(fit, newdata = xy),
nrow = grid.lines, ncol = grid.lines)
fig <- plot_ly(data = gambas) |>
add_trace(
x = ~ comprimento_total,
y = ~ comprimento_cauda,
z = ~ comprimento_cabeca,
type = "scatter3d",
mode = "markers",
opacity = .8
) |>
add_trace(
z = z.pred,
x = x.pred,
y = y.pred,
type = "surface",
opacity = .9
) |>
layout(scene = list(xaxis = list(title = "Total (cm)"),
yaxis = list(title = "Cauda (cm)"),
zaxis = list(title = "Cabeça (mm)"))) |>
hide_legend() |>
hide_colorbar()
fig
```
:::
:::
## O que o R nos diz?
```{r modelo-cabeca-pelo-total-e-cauda}
modelo_linear_multiplo <-
lm(comprimento_cabeca ~ comprimento_total + comprimento_cauda,
data = gambas)
summary(modelo_linear_multiplo)
```
. . .
$$
cabeça = 46.8 + 0.64total - 0.28cauda
$$
## Coeficiente de determinação
* O $R^2$ (coeficiente de determinação) é a proporção da variância na variável
resposta que pode ser explicada pelos preditores
* Varia de 0 a 1
. . .
```{r r-quadrado-modelo-cabeca}
# Extrair o R^2
summary(modelo_linear_multiplo)$r.squared
# Extrair o R^2 ajustado
summary(modelo_linear_multiplo)$adj.r.squared
```
. . .
"O modelo demonstra que `r paste0(format(round(summary(modelo_linear_multiplo)$adj.r.squared * 100, 1), decimal.mark = ","), "%")` da variação no comprimento da cabeça dos gambás pode ser explicado pelo
comprimento total e comprimento da cauda dos gambás."
## Preditores categóricos
#### Preditores com apenas duas categorias
Pergunta de pesquisa: O tamanho das nadadeiras dos pinguins variam conforme
o sexo?
::: columns
::: {.column width="50%"}
```{r boxplot-nadadeira-sexo}
#| echo: false
#| out.width: "100%"
#| fig-align: "center"
dados::pinguins |>
filter(!is.na(sexo)) |>
mutate(
sexo = if_else(sexo == "macho", "Macho", "Fêmea")
) |>
ggplot(aes(y = comprimento_nadadeira, x = sexo, fill = sexo)) +
geom_boxplot(show.legend = FALSE) +
ghibli::scale_fill_ghibli_d("MarnieLight1") +
labs(y = "Comprimento da nadadeira (mm)",
x = "Sexo") +
theme_minimal(20, "Charter")
```
:::
::: {.column width="50%"}
```{r scatterplot-nadadeira-sexo}
#| echo: false
#| out.width: "100%"
#| fig-align: "center"
set.seed(1)
dados::pinguins |>
filter(!is.na(sexo)) |>
mutate(
sexo_macho = if_else(sexo == "macho", 1, 0)
) |>
ggplot(aes(y = comprimento_nadadeira, x = sexo_macho)) +
geom_jitter(width = 0.05, height = 0,
alpha = 0.7, size = 2) +
geom_smooth(method = "lm", se = FALSE,
color = "#0E84B4FF", linewidth = 1.5) +
labs(y = "Comprimento da nadadeira (mm)",
x = "Sexo (macho)") +
scale_x_continuous(breaks = 0:1) +
theme_minimal(20, "Charter")
```
:::
:::
```{r regressao-nadadeira-sexo}
lm(comprimento_nadadeira ~ sexo, data = dados::pinguins)
```
## Preditores categóricos
#### Preditores com 3 ou mais categorias
::: columns
::: {.column width="50%"}
```{r boxplot-nadadeira-especie}
#| echo: false
#| out.width: "100%"
#| fig-align: "center"
dados::pinguins |>
ggplot(aes(y = comprimento_nadadeira, x = especie, fill = especie)) +
geom_boxplot(show.legend = FALSE) +
ghibli::scale_fill_ghibli_d("MononokeMedium", direction = -1) +
labs(y = "Comprimento da nadadeira (mm)",
x = "Espécie") +
theme_minimal(20, "Charter")
```
```{r linear-nadadeira-especie}
lm(comprimento_nadadeira ~ especie, data = dados::pinguins)
```
:::
::: {.column width="50%"}
Modelo:
$$
y_i = \beta_0 + \beta_1x_{1i} + \beta_2x_{2i}
$$
Em que:
$$
x_{1i} = \Bigg \{ \begin{array}{ll} 1 & \text{se for }\texttt{Pinguim-de-barbicha}\\0&\text{caso contrário}\end{array}
$$
$$
x_{2i} = \Bigg \{ \begin{array}{ll} 1 & \text{se for }\texttt{Pinguim-gentoo}\\0&\text{caso contrário}\end{array}
$$
:::
:::
## Heteroscedasticidade
* Inconstância da variabilidade dos erros
. . .
```{r plot-heteroscedasticidade}
#| out.width: "100%"
#| echo: false
#| fig-align: "center"
set.seed(1)
hetero_df <- tibble(
x = runif(50),
y = rnorm(50, mean = x, sd = x * 0.5)
)
cru <- ggplot(hetero_df, aes(x = x, y = y)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
theme_minimal(20, "Charter") +
labs(x = unname(latex2exp::TeX("$x_1$")), y = "Y",
title = unname(latex2exp::TeX("$Y = \\beta_0 + \\beta_1x_1$")))
cru
```
## Lidando com a heteroscedasticidade
* Como diagnosticar?
- Visualização dos resíduos (`gglm::gglm()`)
- Testes (Breuch-Pagan através da função `lmtest::bptest()`)
* Como tratar?
- Transformações na variável resposta ($log(Y)$, $\sqrt{Y}$, $\frac{1}{Y}$)
- Regressão com pesos
. . .
```{r plot-heteroscedasticidade-contornada}
#| out.width: "50%"
#| echo: false
#| fig-align: "center"
raiz <- ggplot(hetero_df, aes(x = x, y = sqrt(y))) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
theme_minimal(20, "Charter") +
labs(x = unname(latex2exp::TeX("$x_1$")),
y = unname(latex2exp::TeX("$\\sqrt{Y}")),
title = unname(latex2exp::TeX("$\\sqrt{Y} = \\beta_0 + \\beta_1x_1$")))
cru + raiz
```
## Multicolinearidade
* Ocorre quando duas ou mais variáveis preditoras são altamente correlacionadas umas com as outras
* Não fornecem informações exclusivas
* Exemplo: preço de um imóvel ($Y$) explicado pela área ($x_1$) e número de cômodos ($x_2$)
. . .
```{r exemplo-multicolinearidade-preco-area-quartos}
#| echo: false
#| out.width: "80%"
#| fig-align: "center"
set.seed(1)
area <- rnorm(100, mean = 1500, sd = 500)
comodos <- round(rnorm(100, mean = 3 + 0.5 * area/100, sd = 1), 0)
preco <- rnorm(100, mean = 200000 + 100 * area + 50000 * comodos, sd = 50000)
area <- area / 10
dados <- data.frame(area, comodos, preco)
ggplot(dados, aes(x = area, y = preco, color = comodos)) +
geom_point() +
scale_color_viridis_c(name = "Cômodos") +
scale_y_continuous(labels = scales::dollar_format(prefix = "R$ ",
big.mark = ".")) +
scale_x_continuous(labels = \(x) unname(latex2exp::TeX(paste0(as.character(x), "$m^2$")))) +
theme_minimal(20, "Charter") +
labs(x = "Área",
y = "Preço do imóvel")
```
## Lidando com a multicolinearidade
::: columns
::: {.column width="50%"}
* Problemas
- Instabilidade numérica
- Desvios padrão inflados
- Interpretação comprometida
* Soluções
- Eliminar uma das variáveis muito correlacionadas
- Consultar o *variance inflation factor* (VIF)
:::
::: {.column width="50%"}
```{r calcular-vif-preco-imovel}
library(car)
regressao_linear <- lm(preco ~ area + comodos,
data = dados)
vif(regressao_linear)
```
* Regra geral
- Valor 1: sem correlação entre o preditor e os demais preditores
- Valor entre 1 e 5: Correlação moderada entre o preditor e demais preditores
- Valor maior que 5: Correlação severa que pode afetar $p$-valores e coeficientes
da regressão, sendo potencialmente não confiáveis
:::
:::
## Outliers
* As observações distantes das outras do conjunto de dados são consideradas **outliers**
. . .
```{r ilustracao-influencia-outlier}
#| echo: false
#| fig-align: "center"
#| out.width: "90%"
set.seed(1)
x <- 1:10
y <- 2*x + rnorm(10, mean = 0, sd = 1)
df <- data.frame(x, y)
# Criando o gráfico sem outlier
p_sem_outlier <- ggplot(df, aes(x = x, y = y)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
scale_x_continuous(limits = c(0, 12.5)) +
scale_y_continuous(limits = c(0, 50)) +
theme_minimal(18, "Charter") +
labs(x = unname(latex2exp::TeX("$x_1$")),
y = unname(latex2exp::TeX("$Y$")))
# Adicionando um outlier aos dados
df[11, ] <- c(12, 50)
# Criando o gráfico com outlier
p_com_outlier <- ggplot(df, aes(x = x, y = y)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
theme_minimal(18, "Charter") +
scale_x_continuous(limits = c(0, 12.5)) +
scale_y_continuous(limits = c(0, 50)) +
labs(x = unname(latex2exp::TeX("$x_1$ com \\textit{outlier}")),
y = NULL)
p_sem_outlier + p_com_outlier
```
## Lidando com outliers
::: columns
::: {.column width="40%"}
* Como diagnosticar?
- Visualização univariada (histograma, boxplot)
- Comparação do valor com o desvio padrão
- Distância de Cook
* Como tratar?
- Transformações $log()$, etc
- Categorização
- Remover valores extremos (não recomendado)
:::
::: {.column width="60%"}
```{r distancia-cook-codigo}
modelo_com_outlier <- lm(y ~ x, data = df)
cooks.distance(modelo_com_outlier)
4 / nrow(df)
```
```{r distancia-cook-com-outlier-plot}
#| echo: false
#| fig-align: "center"
#| out.width: "100%"
modelo_com_outlier <- lm(y ~ x, data = df)
distancia_cook <- cooks.distance(modelo_com_outlier)
df_cook <- data.frame(distancia_cook, row.names = NULL)
ggplot(df_cook, aes(x = as.numeric(row.names(df_cook)), y = distancia_cook)) +
geom_point(size = 3) +
scale_x_continuous(breaks = 1:11) +
geom_hline(yintercept = 4/nrow(df_cook),
color = "darkred", size = 2) +
labs(x = "Observação", y = "Distância de Cook") +
theme_minimal(20, "Charter")
```
:::
:::
## Correlação *vs.* Regressão linear
::: columns
::: {.column width="50%"}
#### Similaridades
* Coeficiente de regressão padronizado é o mesmo que o coeficiente de
correlação de Pearson
* O quadrado do coeficiente de correlação de Pearson é o mesmo que o
$R^2$ na regressão linear simples
* O sinal do coeficiente não-padronizado da regressão será o mesmo do
coeficiente de correlação
* Nenhum dos métodos respondem perguntas de causalidade
:::
::: {.column width="50%"}
#### Diferenças
* A equação da regressão ($\beta_0 + \beta_1x_1$) pode ser usada para
fazer predições de $Y$ baseado em $X$
* Apesar da correlação se referir comumente a relações lineares, o
conceito pode estar associado a outros tipos de associação
* Correlação entre $X$ e $Y$ é a mesma correlação de $Y$ e $X$.
Já na regressão linear, o coeficiente muda
quando comparamos um modelo que prediz $Y$ a partir de $X$ e um modelo
que prediz $X$ a partir de $Y$
:::
:::
## Tarefa de casa
* Ler Capítulo 8 do
[livro OpenIntro Statistics](https://www.openintro.org/book/os/) sobre
*Introduction to linear regression*
- Caso queira se aprofundar, ler Capítulo 9 sobre
*Multiple and logistic regression*
* A partir dos dados `dados_iris` do pacote `dados`,
crie um modelo de
regressão linear simples e um modelo de regressão
linear múltipla
com as variáveis da sua escolha
- Lembre-se que o desfecho deve ser contínuo (numérico)
* Crie um gráfico de dispersão/pontos (*scatterplot*) para acompanhar
sua análise de regressão linear simples