Import de données
Importer des données est souvent l’une des première opérations que l’on effectue lorsque l’on débute sous R, et ce n’est pas la moins compliquée. En cas de problème il ne faut donc pas hésiter à demander de l’aide par les différents moyens disponibles (voir le chapitre Où trouver de l’aide ?) avant de se décourager.
N’hésitez donc pas à relire régulièrement ce chapitre en fonction de vos besoins.
Avant toute chose, il est impératif de bien organiser ses différents fichiers (voir le chapitre dédié). Concernant les données sources que l’on utilisera pour ses analyses, je vous recommande de les placer dans un sous-répertoire dédié de votre projet.
Lorsque l’on importe des données, il est également impératif de vérifier que l’import s’est correctement déroulé (voir la section Inspecter les données du chapitre Premier travail avec les données).
Importer des fichiers texte
Les fichiers texte constituent un des formats les plus largement supportés par la majorité des logiciels statistiques. Presque tous permettent d’exporter des données dans un format texte, y compris les tableurs comme Libre Office, Open Office ou Excel.
Cependant, il existe une grande variétés de format texte, qui peuvent prendre différents noms selon les outils, tels que texte tabulé ou texte (séparateur : tabulation), CSV (pour comma-separated value, sachant que suivant les logiciels le séparateur peut être une virgule ou un point-virgule).
Structure d’un fichier texte
Dès lors, avant d’importer un fichier texte dans R, il est indispensable de regarder comment ce dernier est structuré. Il importe de prendre note des éléments suivants :
- La première ligne contient-elle le nom des variables ? Ici c’est le cas.
- Quel est le caractère séparateur entre les différentes variables (encore appelé séparateur de champs) ? Dans le cadre d’un fichier CSV, il aurait pu s’agir d’une virgule ou d’un point-virgule.
- Quel est le caractère utilisé pour indiquer les décimales (le séparateur décimal) ? Il s’agit en général d’un point (à l’anglo-saxonne) ou d’une virgule (à la française).
- Les valeurs textuelles sont-elles encadrées par des guillemets et, si oui, s’agit-il de guillements simple (
') ou de guillemets doubles (") ? - Pour les variables textuelles, y a-t-il des valeurs manquantes et si oui comment sont-elles indiquées ? Par exemple, le texte
NAest parfois utilisé.
Il ne faut pas hésitez à ouvrir le fichier avec un éditeur de texte pour le regarder de plus près.
Interface graphique avec RStudio
RStudio fournit une interface graphique pour faciliter l’import d’un fichier texte. Pour cela, il suffit d’aller dans le menu File > Import Dataset et de choisir l’option From CSV1. Cette option est également disponible via l’onglet Environment dans le quadrant haut-droite.
Pour la suite de la démonstration, nous allons utiliser le fichier exemple http://larmarange.github.io/analyse-R/data/exemple_texte_tabule.txt.
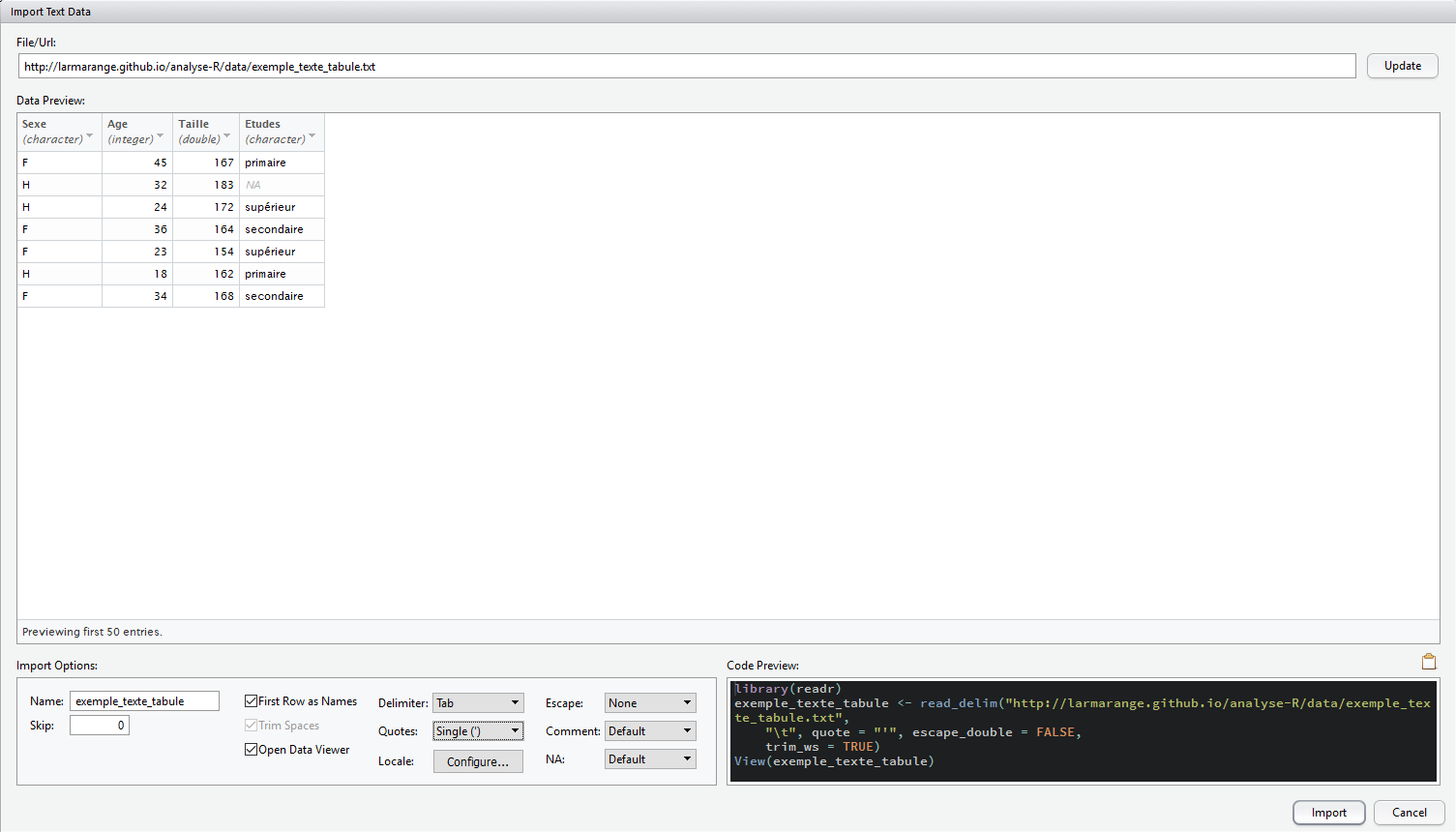
Importer un fichier texte avec RStudio
L’interface de RStudio vous présente sous Import Options les différentes options d’import disponible. La section Data Preview vous permet de voir en temps réel comment les données sont importées. La section Code Preview vous indique le code R correspondant à vos choix. Il n’y a plus qu’à le copier/coller dans un de vos scripts ou à cliquer sur Import pour l’exécuter.
Vous pourrez remarquer que RStudio fait appel à l’extension readr du tidyverse pour l’import des données via la fonction read_csv.
readr essaie de deviner le type de chacune des colonnes, en se basant sur les premières observations. En cliquant sur le nom d’une colonne, il est possible de modifier le type de la variable importée. Il est également possible d’exclure une colonne de l’import (skip).
Dans un script
L’interface graphique de RStudio fournit le code d’import. On peut également l’adapter à ces besoins en consultant la page d’aide de read_csv pour plus de détails. Par exemple :
library(readr)
d <- read_delim("http://larmarange.github.io/analyse-R/data/exemple_texte_tabule.txt",
delim = "\t", quote = "'"
)Rows: 7 Columns: 4
-- Column specification ------------------------------------
Delimiter: "\t"
chr (2): Sexe, Etudes
dbl (1): Age
i Use `spec()` to retrieve the full column specification for this data.
i Specify the column types or set `show_col_types = FALSE` to quiet this message.Le premier élément peut être un lien internet ou bien le chemin local vers un fichier. Afin d’organiser au mieux vos fichiers, voir le chapitre Organiser ses fichiers.
Certains caractères sont parfois précédés d’une barre oblique inversée ou antislash (\). Cela correspond à des caractères spéciaux. En effet, " est utilisé pour délimiter dans le code le début et la fin d’une chaîne de caractères. Comment indiquer à R le caractère " proprement dit. Et bien avec \". De même, \t sera interprété comme une tabulation et non comme la lettre t.
Pour une liste complète des caractères spéciaux, voir ?Quotes.
class(d)[1] "spec_tbl_df" "tbl_df" "tbl" "data.frame" d# A tibble: 7 x 4
Sexe Age Taille Etudes
<chr> <dbl> <dbl> <chr>
1 F 45 167 primaire
2 H 32 183 <NA>
3 H 24 172 supérieur
4 F 36 164 secondaire
5 F 23 154 supérieur
6 H 18 162 primaire
7 F 34 168 secondaireL’objet renvoyé est un tableau de données ou data.frame. Plus précisément, il s’agit d’un tibble, c’est-à-dire un tableau de données légèrement amélioré facilement utilisable avec les différentes extensions du tidyverse. Pas de panique, c’est un tableau de données comme les autres. Disons qu’il est possible de faire un peu plus de choses avec. Pour cela, voir le chapitre dédié à dplyr.
readr propose plusieurs fonctions proches : read_delim, read_csv, read_csv2 et read_tsv. Elles fonctionnent toutes de manière identique et ont les mêmes arguments. Seule différence, les valeurs par défaut de certainsparamètres.
Dans des manuels ou des exemples en ligne, vous trouverez parfois mention des fonctions read.table, read.csv, read.csv2, read.delim ou encore read.delim2. Il s’agit des fonctions natives et historiques de R (extension utils) dédiées à l’import de fichiers textes. Elles sont similaires à celles de readr dans l’idée générale mais diffèrent dans leurs détails et les traitements effectués sur les données (pas de détection des dates par exemple). Pour plus d’information, vous pouvez vous référer à la page d’aide de ces fonctions.
Importer depuis des logiciels de statistique
Plusieurs extensions existent pour importer des fichiers de données issus d’autres logiciels de statistiques. En premier lieu, il y a foreign, installée par défaut avec R et décrite en détails dans le manuel R Data Import/Export disponible sur http://cran.r-project.org/manuals.html. Un des soucis majeurs de cette extension réside dans la manière dont elle traite les métadonnées utilisées en particulier dans les fichiers SAS, SPSS et Stata, à savoir les étiquettes de variable, les étiquettes de valeur et les valeurs manquantes
déclarées. En effet, chaque fonction va importer ces métadonnées sous la forme d’attributs dont le nom diffère d’une fonction à l’autre. Par ailleurs, selon les options retenues, les variables labellisées seront parfois transformées ou non en facteurs. Enfin, foreign ne sait pas toujours importer les différents types de variables représentant des dates et des heures.
L’extension haven (qui fait partie du tidyverse
) tente de remédier à plusieurs des limitations rencontrées avec foreign :
- le format des métadonnées importé est uniforme, quel que soit le type de fichier source (SAS, SPSS ou Stata) ;
- les variables labellisées ne sont pas transformées en facteurs, mais héritent d’une nouvelle classe
haven_labelled, la valeur initiale restant inchangée ; - les différents formats de date sont convertis dans des classes R appropriées, utilisables en particulier avec
lubridate; havenpeut lire les fichiers SAS natifs (extension.sas7bdat) ce que ne peut pas faireforeign;havenpeut lire les fichiers Stata 13 et 14, alors queforeignne sait lire ces fichiers que jusqu’à la version 12 ;- les tableaux de données produits ont directement la classe
tbl_dfce qui permets d’utiliser directement les fonctionnalités de l’extensiondplyr.
À noter, il est également possible d’utiliser l’interface graphique de RStudio pour l’import des fichiers SPSS, Stata, SAS et Excel.
Données labellisées
À la différence de foreign, haven ne convertit pas les variables avec des étiquettes de valeurs en facteurs mais en vecteurs labellisés du type haven_labelled qui sont présentés en détail dans le chapitre Facteurs et vecteurs labellisés et dans le chapitre recodage de variables où est discutée la question de savoir à quel moment convertir les données labellisées.
SPSS
Les fichiers générés par SPSS sont de deux types : les fichiers SPSS natifs natifs (extension .sav) et les fichiers au format SPSS export (extension .por).
Dans les deux cas, on aura recours à la fonction read_spss :
library(haven)
donnees <- read_spss("data/fichier.sav", user_na = TRUE)Gestion des valeurs manquantes
Dans SPSS, il est possible de définir des valeurs à considérées comme manquantes. Plus précisément jusqu’à 3 valeurs spécfiques et/ou les valeurs comprises entre un minimum et un maximum. Par défaut, read_spss convertir toutes ces valeurs en NA lors de l’import.
Or, il est parfois important de garder les différentes valeurs originelles, notamment dans le cadre de l’analyse de données d’enquête, un manquant du type ne sait pas
n’étant pas équivalent à un manquant du type refus
ou du type variable non collectée
.
Dès lors, nous vous recommandons d’appeler read_spss avec l’option user_na = TRUE. Dans ce cas-là, les valeurs manquantes définies dans SPSS ne seront pas converties en NA, tout en conservant la définition des valeurs définies comme manquantes. Il sera alors toujours possible de convertir, dans un second temps et en fonction des besoins, ces valeurs à considérer comme manquantes en NA grace aux fonctions de l’extension labelled, en particulier user_na_to_na, na_values et na_range.
À noter que les fonctions describe et freq de l’extension questionr que nous arboderons dans d’autres chapitres savent exploiter ces valeurs à considérer comme manquantes.
Si vous préférez utiliser l’extension foreign, la fonction correspondante est read.spss. On indiquera à la fonction de renvoyer un tableau de données avec l’argument to.data.frame = TRUE.
Par défaut, les variables numériques pour lesquelles des étiquettes de valeurs ont été définies sont transformées en variables de type facteur, les étiquettes définies dans SPSS étant utilisées comme labels du facteur. De même, si des valeurs manquantes ont été définies dans SPSS, ces dernières seront toutes transformées en NA (R ne permettant pas de gérer plusieurs types de valeurs manquantes). Ce comportement peut être modifié avec use.value.labels et use.missings.
library(foreign)
donnees <- read.spss("data/fichier.sav", to.data.frame = TRUE, use.value.labels = FALSE, use.missings = FALSE)Il est important de noter que read.spss de l’extension foreign ne sait pas importer les dates. Ces dernières sont donc automatiquement transformées en valeurs numériques.
SPSS stocke les dates sous la forme du nombre de secondes depuis le début du calendrier grégorien, à savoir le 14 octobre 1582. Dès lors, si l’on des dates dans un fichier SPSS et que ces dernières ont été converties en valeurs numériques, on pourra essayer la commande suivante :
donnees$date <- as.POSIXlt(donnees$date, origin = "1582-10-14")SAS
Les fichiers SAS se présentent en général sous deux format : format SAS export (extension .xport ou .xpt) ou format SAS natif (extension .sas7bdat).
Les fichiers SAS natifs peuvent être importées directement avec read_sas de l’extension haven :
library(haven)
donnees <- read_sas("data/fichier.sas7bdat")Au besoin, on pourra préciser en deuxième argument le nom d’un fichier SAS catalogue (extension .sas7bcat) contenant les métadonnées du fichier de données.
library(haven)
donnees <- read_sas("data/fichier.sas7bdat", "data/fichier.sas7bcat")Les fichiers au format SAS export peuvent être importés via la fonction read.xport de l’extension foreign. Celle-ci s’utilise très simplement, en lui passant le nom du fichier en argument :
library(foreign)
donnees <- read.xport("data/fichier.xpt")Stata
Pour les fichiers Stata (extension .dta), on aura recours aux fonctions read_dta et read_stata de l’extension haven. Ces deux fonctions sont identiques.
library(haven)
donnees <- read_dta("data/fichier.dta")Gestion des valeurs manquantes
Dans Stata, il est possible de définir plusieurs types de valeurs manquantes, qui sont notées sous la forme .a à .z. Pour conserver cette information lors de l’import, haven a introduit dans R le concept de tagged NA ou tagged missing value. Plus de détails sur ces données manquantes étiquettées
, on se référera à la page d’aide de la fonction tagged_na.
Si l’on préfère utiliser l’extension foreign, on aura recours à la fonction read.dta.
L’option convert.factors indique si les variables labellisées doit être converties automatiquement en facteurs. Pour un résultat similaire à celui de haven, on choisira donc :
library(foreign)
donnees <- read.dta("data/fichier.dta", convert.factors = FALSE)L’option convert.dates permet de convertir les dates du format Stata dans un format de dates géré par R. Cependant, cela ne marche pas toujours. Dans ces cas là, l’opération suivante peut fonctionner. Sans garantie néanmoins, il est toujours vivement conseillé de vérifier le résultat obtenu !
donnees$date <- as.Date(donnees$Date, origin = "1960-01-01")Excel
Une première approche pour importer des données Excel dans R consiste à les exporter depuis Excel dans un fichier texte (texte tabulé ou CSV) puis de suivre la procédure d’importation d’un fichier texte.
Une feuille Excel peut également être importée directement avec l’extension readxl qui appartient à la même famille que haven et readr.
La fonction read_excel permet d’importer à la fois des fichiers .xls (Excel 2003 et précédents) et .xlsx (Excel 2007 et suivants).
library(readxl)
donnees <- read_excel("data/fichier.xlsx")Une seule feuille de calculs peut être importée à la fois. On pourra préciser la feuille désirée avec sheet en indiquant soit le nom de la feuille, soit sa position (première, seconde, …).
donnees <- read_excel("data/fichier.xlsx", sheet = 3)
donnees <- read_excel("data/fichier.xlsx", sheet = "mes_donnees")On pourra préciser avec col_names si la première ligne contient le nom des variables.
Par défaut, read_excel va essayer de deviner le type (numérique, textuelle, date) de chaque colonne. Au besoin, on pourra indiquer le type souhaité de chaque colonne avec col_types.
RStudio propose également pour les fichiers Excel un assitant d’importation, similaire à celui pour les fichiers texte, permettant de faciliter l’import.
Une première alternative est l’extenstion openxlsx et sa fonction read.xlsx.
Une seconde alternative est l’extension xlsx qui propose deux fonctions différentes pour importer des fichiers Excel : read.xlsx et read.xlsx2. La finalité est la même mais leur fonctionnement interne est différent. En cas de difficultés d’import, on pourra tester l’autre. Il est impératif de spécifier la position de la feuille de calculs que l’on souhaite importer.
library(xlsx)
donnees <- read.xlsx("data/fichier.xlsx", 1)dBase
L’Insee et d’autres producteur de données diffusent leurs fichiers au format dBase (extension .dbf). Ceux-ci sont directement lisibles dans R avec la fonction read.dbf de l’extension foreign.
library(foreign)
donnees <- read.dbf("data/fichier.dbf")La principale limitation des fichiers dBase est de ne pas gérer plus de 256 colonnes. Les tables des enquêtes de l’Insee sont donc parfois découpées en plusieurs fichiers .dbf qu’il convient de fusionner avec la fonction merge. L’utilisation de cette fonction est détaillée dans le chapitre sur la fusion de tables.
Données spatiales
Shapefile
Les fichiers Shapefile sont couramment utilisés pour échanger des données géoréférencées. La majorité des logiciels de SIG (systèmes d’informations géographiques) sont en capacité d’importer et d’exporter des données dans ce format.
Un shapefile contient toute l’information liée à la géométrie des objets décrits, qui peuvent être :
- des points
- des lignes
- des polygones
Son extension est classiquement .shp et il est toujours accompagné de deux autres fichiers de même nom et d’extensions :
- un fichier
.dbf, qui contient les données attributaires relatives aux objets contenus dans le shapefile - un fichier
.shx, qui stocke l’index de la géométrie
D’autres fichiers peuvent être également fournis :
.sbnet.sbx- index spatial des formes..fbnet.fbx- index spatial des formes pour les shapefile en lecture seule.ainet.aih- index des attributs des champs actifs dans une table ou dans une table d’attributs du thème.prj- information sur le système de coordonnées.shp.xml- métadonnées du shapefile..atx- fichier d’index des attributs pour le fichier.dbf.qix
En premier lieu, il importe que tous les fichiers qui compose un même shapefile soit situés dans le même répertoire et aient le même nom (seule l’extension étant différente).
L’extension maptools fournit les fonctions permettant d’importer un shapefile dans R. Le résultat obtenu utilisera l’une des différentes classes spatiales fournies par l’extension sp.
La fonction générique d’import est readShapeSpatial :
library(maptools)
donnees_spatiales <- readShapeSpatial("data/fichier.shp")Si l’on connait déjà le type de données du shapefile (points, lignes ou polygones), on pourra utiliser directement readShapePoints, readShapeLines ou readShapePoly.
Rasters
Il existe de multiples formats pour stocker des données matricielles spatiales. L’un des plus communs est le format ASCII grid aussi connu sous le nom de Arc/Info ASCII grid ou ESRI grid. L’extension de ce format n’est pas toujours uniforme. On trouve parfois .asc ou encore .ag voir même .txt.
Pour importer ce type de fichier, on pourra avoir recours à la fonction readAsciiGrid de l’extension maptools. Le résultat sera, par défaut, au format SpatialGridDataFrame de l’extension sp.
library(maptools)
donnees_spatiales <- readAsciiGrid("data/fichier.asc")L’extension raster permet d’effectuer de multiples manipulations sur les données du type raster. Elle est en capacité d’importer des données depuis différents formats (plus précisément les formats pris en charge par la librairie GDAL, http://www.gdal.org/).
De plus, les fichiers raster pouvant être particulièrement volumineux (jusqu’à plusieurs Go de données), l’extension raster est capable de travailler sur un fichier raster sans avoir à le charger intégralement en mémoire.
Pour plus d’informations, voir les fonctions raster et getValues.
Connexion à des bases de données
Interfaçage via l’extension DBI
R est capable de s’interfacer avec différents systèmes de bases de données relationnelles, dont SQLite, MS SQL Server, PostgreSQL, MariaDB, etc.
Pour illustrer rapidement l’utilisation de bases de données, on va créer une base SQLite d’exemple à l’aide du code R suivant, qui copie la table du jeu de données mtcars dans une base de données bdd.sqlite :
[1] TRUElibrary(DBI)
library(RSQLite)
con <- DBI::dbConnect(RSQLite::SQLite(), dbname = "bdd.sqlite")
data(mtcars)
mtcars$name <- rownames(mtcars)
dbWriteTable(con, "mtcars", mtcars)
dbDisconnect(con)Si on souhaite se connecter à cette base de données par la suite, on peut utiliser l’extension DBI, qui propose une interface générique entre **R// et différents systèmes de bases de données. On doit aussi avoir installé et chargé l’extension spécifique à notre base, ici RSQLite. On commence par ouvrir une connexion à l’aide de la fonction dbConnect de DBI :
library(DBI)
library(RSQLite)
con <- DBI::dbConnect(RSQLite::SQLite(), dbname = "bdd.sqlite")La connexion est stockée dans un objet con, qu’on va utiliser à chaque fois qu’on voudra interroger la base.
On peut vérifier la liste des tables présentes et les champs de ces tables avec dbListTables et dbListFields :
dbListTables(con)[1] "mtcars"dbListFields(con, "mtcars") [1] "mpg" "cyl" "disp" "hp" "drat" "wt" "qsec" "vs"
[9] "am" "gear" "carb" "name"On peut également lire le contenu d’une table dans un objet de notre environnement avec dbReadTable :
cars <- dbReadTable(con, "mtcars")On peut également envoyer une requête SQL directement à la base et récupérer le résultat avec dbGetQuery :
dbGetQuery(con, "SELECT * FROM mtcars WHERE cyl = 4") mpg cyl disp hp drat wt qsec vs am gear carb
1 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
2 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
3 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
4 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
5 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
6 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
7 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
8 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
9 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
10 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
11 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2
name
1 Datsun 710
2 Merc 240D
3 Merc 230
4 Fiat 128
5 Honda Civic
6 Toyota Corolla
7 Toyota Corona
8 Fiat X1-9
9 Porsche 914-2
10 Lotus Europa
11 Volvo 142EEnfin, quand on a terminé, on peut se déconnecter à l’aide de dbDisconnect :
dbDisconnect(con)Ceci n’est évidemment qu’un tout petit aperçu des fonctionnalités de DBI.
Utilisation de dplyr et dbplyr
L’extension dplyr est dédiée à la manipulation de données, elle est présentée dans un chapitre dédié. En installant l’extension complémentaire dbplyr, on peut utiliser dplyr directement sur une connection à une base de données générée par DBI :
library(DBI)
library(RSQLite)
library(dplyr)
con <- DBI::dbConnect(RSQLite::SQLite(), dbname = "bdd.sqlite")La fonction tbl notamment permet de créer un nouvel objet qui représente une table de la base de données :
cars_tbl <- tbl(con, "mtcars")Ici l’objet cars_tbl n’est pas un tableau de données, c’est juste un objet permettant d’interroger la table de notre base de données.
On peut utiliser cet objet avec les verbes de dplyr :
cars_tbl %>%
filter(cyl == 4) %>%
select(name, mpg, cyl)dbplyr s’occupe, de manière transparente, de transformer les instructions dplyr en requête SQL, d’interroger la base de données et de renvoyer le résultat. De plus, tout est fait pour qu’un minimum d’opérations sur la base, parfois coûteuses en temps de calcul, ne soient effectuées.
Il est possible de modifier des objets de type tbl, par exemple avec mutate :
cars_tbl <- cars_tbl %>% mutate(type = "voiture")
Dans ce cas la nouvelle colonne type est bien créée et on peut y accéder par la suite. Mais cette création se fait dans une table temporaire : elle n’existe que le temps de la connexion à la base de données. À la prochaine connexion, cette nouvelle colonne n’apparaîtra pas dans la table.
Bien souvent on utilisera une base de données quand les données sont trop volumineuses pour être gérées par un ordinateur de bureau. Mais si les données ne sont pas trop importantes, il sera toujours plus rapide de récupérer l’intégralité de la table dans notre session R pour pouvoir la manipuler comme les tableaux de données habituels. Ceci se fait grâce à la fonction collect de dplyr :
cars <- cars_tbl %>% collect()Ici, cars est bien un tableau de données classique, copie de la table de la base au moment du collect.
Et dans tous les cas, on n’oubliera pas de se déconnecter avec :
dbDisconnect(con)Ressources
Pour plus d’informations, voir la documentation très complète (en anglais) proposée par RStudio.
Par ailleurs, depuis la version 1.1, RStudio facilite la connexion à certaines bases de données grâce à l’onglet Connections. Pour plus d’informations on pourra se référer à l’article (en anglais) Using RStudio Connections.
Autres sources
R offre de très nombreuses autres possibilités pour accéder aux données. Il est ainsi possible d’importer des données depuis d’autres applications qui n’ont pas été évoquées (Epi Info, S-Plus, etc.), de lire des données via ODBC ou des connexions réseau, etc.
Pour plus d’informations on consultera le manuel R Data Import/Export :
http://cran.r-project.org/manuals.html.
La section Database Management du site Awesome R fournit également une liste d’extensions permettant de s’interfacer avec différents gestionnaires de bases de données.
Sauver ses données
R dispose également de son propre format pour sauvegarder et échanger des données. On peut sauver n’importe quel objet créé avec R et il est possible de sauver plusieurs objets dans un même fichier. L’usage est d’utiliser l’extension .RData pour les fichiers de données R. La fonction à utiliser s’appelle tout simplement save.
Par exemple, si l’on souhaite sauvegarder son tableau de données d ainsi que les objets tailles et poids dans un fichier export.RData :
save(d, tailles, poids, file = "export.RData")À tout moment, il sera toujours possible de recharger ces données en mémoire à l’aide de la fonction load :
load("export.RData")Si entre temps vous aviez modifié votre tableau d, vos modifications seront perdues. En effet, si lors du chargement de données, un objet du même nom existe en mémoire, ce dernier sera remplacé par l’objet importé.
La fonction save.image est un raccourci pour sauvergarder tous les objets de la session de travail dans le fichier .RData (un fichier un peu étrange car il n’a pas de nom mais juste une extension). Lors de la fermeture de RStudio, il vous sera demandé si vous souhaitez enregistrer votre session. Si vous répondez Oui, c’est cette fonction save.image qui sera appliquée.
save.image()[1] FALSEL’option CSV fonctionne pour tous les fichiers de type texte, même si votre fichier a une autre extension,
.txtpar exemple↩︎
